作者:北京师范大学 龙宇航,[email protected]
代码来源(见本页底部):周思远
在使用wtc计算脑间神经同步后,我们需要在多个频率段、多个通道组合上对神经同步值进行统计检验,因此当进行频段选择时,面临多重比较的问题。为了解决多重比较的问题,可以采取基于参数或非参数检验的多重比较矫正的方法。由于基于非参数检验的多重比较矫正对数据的分布形态没有严格要求,因此具有更广泛的应用场景 (Maris and Oostenveld, 2007)。本文即介绍基于随机置换的非参数检验的方法 (Zheng et al., 2020; Long et al., 2021)。
在寻找感兴趣的效应时,我们采取了基于频率簇(Cluster)的方法,即在频率方向寻找连续显著的Cluster,该方法比基于最强效应点的方法具有更为优秀的抗噪音能力。值得注意的是,我们并没有沿着通道的方向去寻找连续显著的通道簇,这是因为沿着通道方向寻找Cluster容易受到生理噪音的影响。
下面进入具体的实操部分。假设本例招募了22对组1被试及22对组2被试,每对被试分别进行3种条件的任务,因此本例是2(组别,被试间因素)*3(条件,被试内因素)的实验设计。本例对神经同步值进行2*3的混合方差分析,并关注交互作用。
具体来讲,进行置换检验需要进行以下几个步骤:1. 重采样;2. 对随机样本进行计算及统计;3. 计算真实样本的统计量;4. 真实样本与随机样本的对比。下面依次进行介绍。
1. 重采样
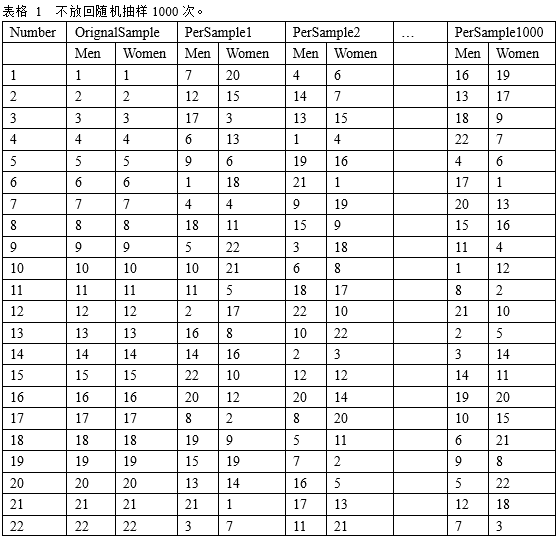
首先,在每种实验条件下都进行重采样。置换检验采取的是不放回抽样,一般来讲至少需要1000个随机样本(不放回随机采样)。以组1在条件1下的重采样为例,本例中的每对被试由一男一女组成,表1中Original Sample (OS) 指组1在条件1时的配对情况,即原始配对。而每个Permutation Sample (PS) 中的22对被试并不是真实被试,如在PS1的第一对被试中,第7号男性在条件1下的血氧信号与第20号女性在条件1下的血氧信号进行了匹配,因此并不是真实互动条件下的神经信号。同样地,对每个水平都进行重采样。
2. 对随机样本进行计算及统计
2.1 对每个随机配对进行WTC的计算得到神经同步值。
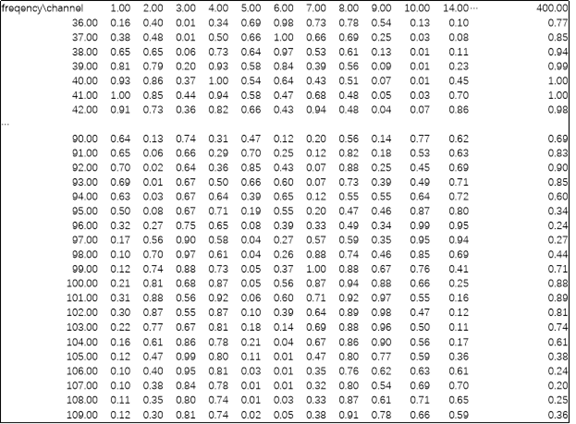
2.2 对每个随机样本(PS)的神经同步值进行所关心的统计分析。在本例中我们得到交互作用的P值矩阵,其中一个维度为频率,一个维度为通道。假设第1个随机样本我们得到了以下统计P值矩阵(表2),表中显示有400个通道,以及109个频率点。
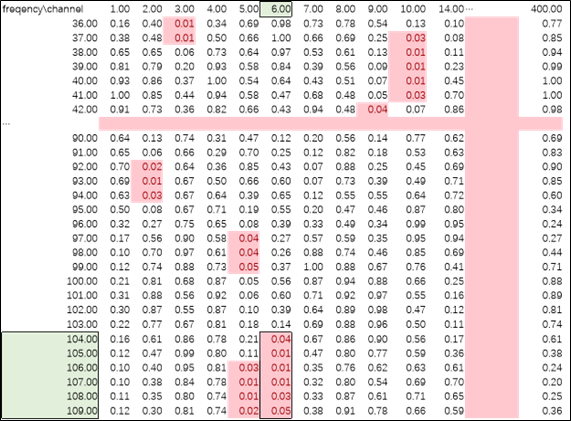
2.3 按频率方向挑选最长连续的P < 0.05的Cluster,得到该Cluster所对应的频段以及通道。具体来讲,在表3中标红的为P < 0.05的值,我们可以看到被框起的红色部分为表中按频率方向(纵向)最长的Cluster,该Cluster所对应的频率段为104 – 109,通道组合为6。至此,我们已经找到了该随机样本最长的Cluster及其所对应的统计值、对应的通道、以及对应的频率段。寻找最长连续P < 0.05 Cluster的代码见本页底部。
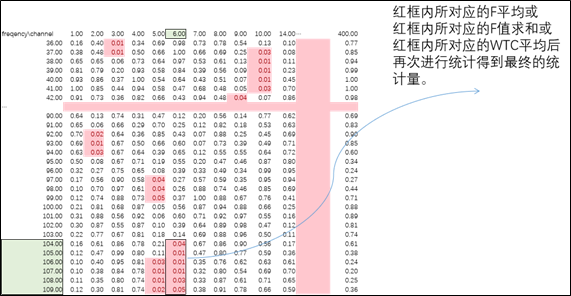
2.4 得到最终用于画图的统计值。有3种方法可以得到该值,如下图,第一种为将该Cluster所对应的F值平均。第二种为将该Cluster所对应的F值求和。第三种为将该Cluster所对应的频段以及通道的WTC值进行平均,对平均后的WTC值再次进行统计分析。
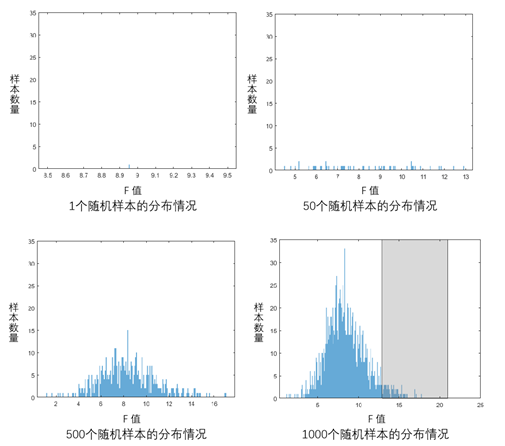
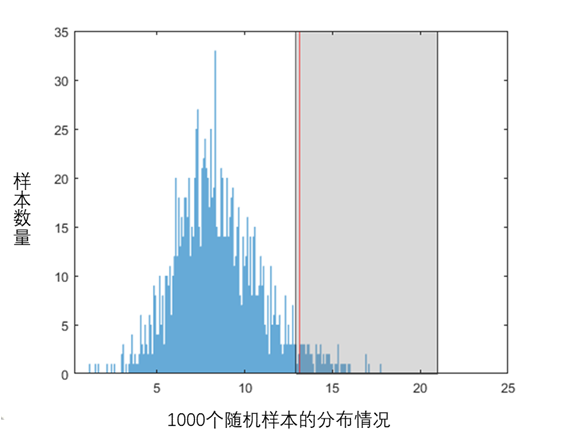
2.5 对每个随机样本(PS)都进行上述2.1-2.4的操作,最终得到1000个随机样本(PS)统计值的95百分位数,如下图(图中为示意图,统计值与上表并不对应),图中横轴代表最终用于画图的F值,纵轴代表样本数量。在计算完第一个随机样本后,我们发现其F值为8.97(图2中左上角子图);依此类推,当计算完1000个随机样本(PS)后,我们能够画出1000个随机样本的分布情况(图2中右下角子图),并根据该分布得到大于95百分位数区域(灰色区域)。
3. 计算真实样本的统计量
3.1 对真实样本进行WTC计算。
3.2 对真实样本的神经同步值进行统计分析得到二维的统计值矩阵(一个维度为频率一个维度为通道)。
3.3 按频率方向挑选所有连续的P < 0.05的Cluster所对应的频段以及通道。
3.4 将每一个符合要求的Cluster所对应的F值进行平均或求和或对应的WTC值进行平均后再次进行统计分析。
4. 真实样本与随机样本的对比
看真实样本中哪个cluster能够落在灰色区域。在本例中,红色线条为真实样本的某一个Cluster所对应的F值,落在了灰色区域内,表明在0.05水平显著。至此,我们已基于频率簇(Cluster)的置换检验(Permutation)方法选取出了感兴趣的频段。
参考文献:
1. Long Y, Zheng L, Zhao H, Zhou S, Zhai Y, Lu C (2021) Interpersonal Neural Synchronization during Interpersonal Touch Underlies Affiliative Pair Bonding between Romantic Couples. Cerebral cortex 31:1647-1659.
2. Maris E, Oostenveld R (2007) Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods 164:177-190.
3. Zheng L, Liu W, Long Y, Zhai Y, Zhao H, Bai X, Zhou S, Li K, Zhang H, Liu L, Guo T, Ding G, Lu C (2020) Affiliative bonding between teachers and students through interpersonal synchronisation in brain activity. Social cognitive and affective neuroscience 15:97-109.
MatLab 代码:
function T_selected = bandSelect(pmap, threshold_peak, threshold_bound)
% Dependency: findContineElement
% Input: pmap: the p-value map (fs x ch)
% threshold_peak: the threshold of the peak point
% threshold_bound: the threshold to determine adjacent points around
% peak point
% Output: T_selected: first column is channel pair ID, second column is fsband
% By Siyuan Zhou,2020/10
% Bug Fix: 2021/3
[t2_pos1,t2_pos2]=find(pmap<=threshold_peak);
source_x = t2_pos2;
source_y = t2_pos1;
selected = {};
value = {};
for ii = 1:size(source_x)
pos0 = source_y(ii);
raw = pmap(:,source_x(ii));
select_chwise = findContinueElement(raw, pos0, threshold_bound);
selected{ii}=select_chwise;
value{ii} = raw(select_chwise);
end
T = table(source_x,selected',value','VariableNames',{'ch' 'fs' 'value'});
% convert fs and value to string to remove duplicated data
tempT = T(:,{'ch','fs'});
tempT.fs = cellfun(@num2str,T.fs,'UniformOutput',false);
[B, ci]=unique(tempT(:,[1 2]));
T_selected= T(ci,:);
function continual_set = findContinueElement(raw, pos0, threshold)
% find continual elements in a vector based on a certain threshold
% Input: raw: the vector to find continual elements
% pos0: the start position
% threshold: to determine whether an element belongs to the continual vector
% Output: continual_set: the positions of continual elements
% By Siyuan Zhou, 2020/10
pos = pos0;
idx = 1;
pos_idx = [];
while(pos >0 & pos<=numel(raw) & raw(pos)<threshold)
pos_idx(idx) = pos;
pos = pos +1;
idx = idx+1;
end
pos = pos0 - 1;
while(pos >0 & pos<=numel(raw) & raw(pos)<threshold)
pos_idx(idx) = pos;
pos = pos -1;
idx = idx+1;
end
continual_set = sort(pos_idx);
end
end